中国科学技术大学生命科学与医学部刘海燕教授、陈泉副教授团队与信息科学技术学院李厚强教授团队合作,开发了一种基于深度学习为给定主链结构从头设计氨基酸序列的算法ABACUS-R,在实验验证中,ABACUS-R的设计成功率和设计精度超过了原有统计能量模型ABACUS。相关成果以“Rotamer-Free Protein Sequence Design Based on Deep Learning and Self-Consistency”为题于北京时间2022年7月21日发表于Nature Computational Science。
刘海燕教授、陈泉副教授团队致力于发展数据驱动的蛋白质设计方法,建立并实验验证了利用神经网络能量函数从头设计主链结构的SCUBA模型,以及对给定主链结构设计氨基酸序列的统计能量函数ABACUS。然而,通过优化能量函数来进行序列设计的方法在成功率、计算效率等方面仍有不足。近期有多项研究表明,用深度学习进行氨基酸序列设计能够在天然氨基酸残基类型恢复率等计算指标上超过能量函数方法;但截至目前已正式发表的工作中,对相关方法的实验验证结果远未达到能量函数方法的成功率。该论文报道的ABACUS-R模型,则不仅在计算指标上超过ABACUS,在实验验证中成功率和结构精度也有大幅提高。
用ABACUS-R进行序列设计的方法由两部分组成(图1)。第一部分为预训练的编码器-解码器网络:该网络用Transformer把中心氨基酸残基的化学和空间结构环境映射为隐空间表示向量,再用多层感知机网络将该向量解码为包括中心残基氨基酸类型在内的多种真实特征(图1a)。在方法的第二部分,经用非冗余天然蛋白序列结构数据训练后,ABACUS-R编码器-解码器被用于给定主链结构的全部或部分氨基酸序列从头设计。具体为:从任意初始序列出发,对各个类型待定残基分别应用ABACUS-R编码器-解码器,得到环境依赖的最适宜残基类型,并反复迭代至不同位点的残基类型最大程度自洽(图1b)。
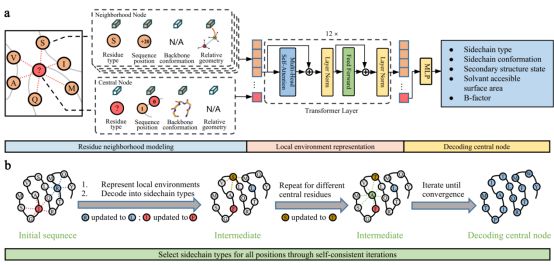
图1. 用ABACUS-R模型进行蛋白质序列设计的原理。(a) 预训练的编码器-解码器网络;(b)采用自洽迭代策略进行全序列从头设计。
在理论验证的基础上,中国科大团队尝试了实验表征用ABACUS-R对3个天然主链结构重新设计的57条序列;其中86%的序列(49条)可溶表达并能折叠为稳定单体;实验解析的5个高分辨晶体结构与目标结构高度一致(主链原子位置均方根位移在1Å以下)(图2)。此外,与以前报道的从头设计蛋白相似,ABACUS-R从头设计的蛋白表现出超高热稳定性,去折叠温度大多可达100℃以上。
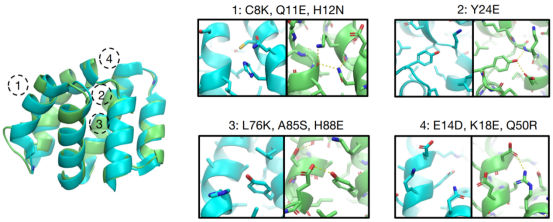
图2. 左侧图为实验验证采用的一个目标主链结构(天蓝色)与相应ABACUS-R设计蛋白晶体结构(绿色)的叠合比较。在右侧展示的局部结构放大图中,ABACUS-R设计蛋白的残基间氢键等极性相互作用不同于天然结构。
相较于ABACUS模型,ABACUS-R序列设计更高的成功率和结构精度进一步增强了数据驱动蛋白质从头设计方法的实用性。ABACUS-R还提供了一种对蛋白质局部结构信息的预训练表示方式,可用于序列设计以外的其他任务。
我校生命科学与医学部刘海燕教授、陈泉副教授、信息科学技术学院李厚强教授为该论文通讯作者。生命科学与医学部硕士生刘宇枫、博士生张璐、信息科学技术学院博士生王炜伦为该论文共同第一作者。该研究工作得到了科技部、国家自然科学基金委和中国科学院的资助支持。
原文链接:https://www.nature.com/articles/s43588-022-00273-6
(生命科学与医学部、信息科学技术学院、微尺度国家研究中心、细胞动力学教育部重点实验室、科研部)
