组蛋白去乙酰化酶(Histone deacetylases, HDACs)是一种进化上保守的酶,可以去除组蛋白的乙酰修饰,在表观遗传基因沉默中发挥核心作用。I类 HDAC是癌症、炎症、感染和神经系统疾病的表观遗传治疗的重要靶点,H3K18Ac是癌症进展的重要标志,也是抗癌治疗的潜在靶点;但催化H3K18Ac去乙酰化的HDAC酶和机制尚不清楚。
中国科大生命科学与医学部王雪娟、蔡刚教授团队合作近期在Cell Research期刊上在线发表了题为Structural basis for nucleosome binding and catalysis by the yeast Rpd3S/HDAC holoenzyme的研究论文。该研究解析了酵母Rpd3S/HDAC全酶复合体结合特定修饰核小体底物的复合物结构,首次发现完整的天然底物H3尾巴(1-24 aa)结合在Rpd3S底物结合口袋,且H3K18残基的侧链朝向催化中心待催化;阐明了Rpd3S底物特异性的结构基础以及通过构象变化实现多位点催化的机制,尤其是Rco1亚基在核小体结合和位点特异性催化中起着关键作用,有望为研制高特异性Rpd3S/Sin3B抑制剂用于癌症治疗提供新靶点。

王雪娟和蔡刚的研究团队首先通过大规模培养酵母细胞和内源性蛋白纯化获得高度均一的Rpd3S复合体全酶,优化并组装了最佳单核小体底物(H3K36me3修饰;仅在核小体一端伸出70bp linker DNA)。在不引入任何化学交联剂干扰的情况下,成功体外组装了Rpd3S-核小体复合物,解析了3.7 Å分辨率的复合物结构(图1)。
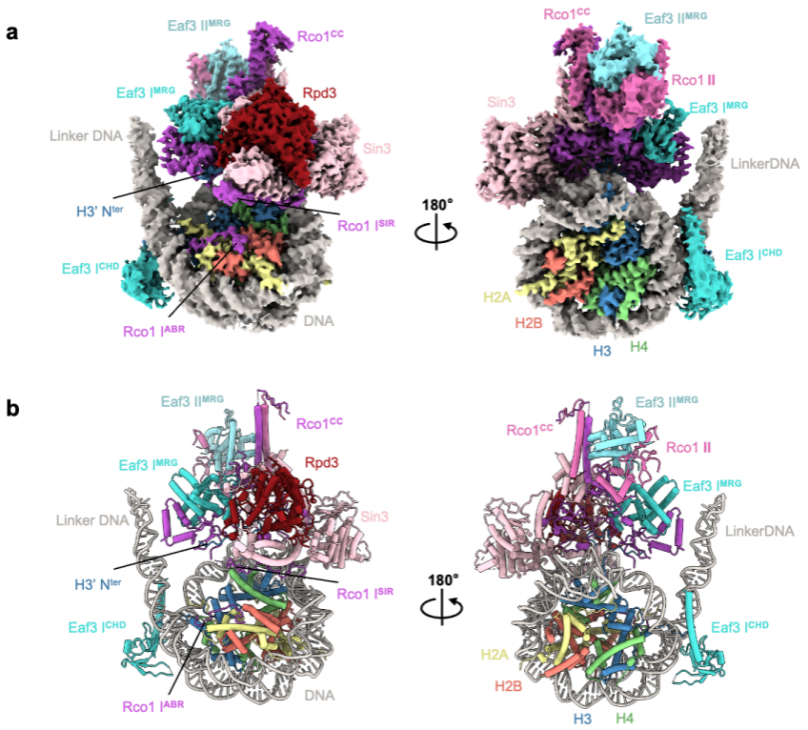
图1. Rpd3S结合H3K36me3修饰的核小体的整体结构
该结构揭示了Rpd3S包含两个Rco1和两个Eaf3拷贝,它们通过Rco1 C-端卷曲区域(CC)进行二聚化,Eaf3 CHD结构域识别H3K36me3标记并与核小体DNA相互作用。 Sin3作为支架蛋白和Rco1亚基一起协调了复合物的组装和催化亚基Rpd3的包裹;同时,Sin3-nucleosomeDNA、Rco1-nucleosomelinker DNA结合界面共同帮助引导Rpd3S精准锚定在核小体底物上。
Rco1 N端的ABR结构域通过R61和K64残基直接锚定在组蛋白的酸性斑块的表面,并通过R50和R51残基在SHL-6.5处与nucleosomeDNA结合。此外,Rco1 PHD1结构域的D261侧链直接与H3K4me0相互作用、参与底物的识别;而Rco1 PHD2结构域与Rpd3相互作用参与复合物整体结构的组装和稳定。
H3K18Ac是癌症进展的重要标志,也是抗癌治疗的潜在靶点。该结构首次发现完整的天然底物H3尾巴(1-24 aa)结合在Rpd3S底物结合口袋,并且H3K18残基的侧链朝向催化中心。这些结构上的重要发现,被体外的功能实验进一步地得到证实。
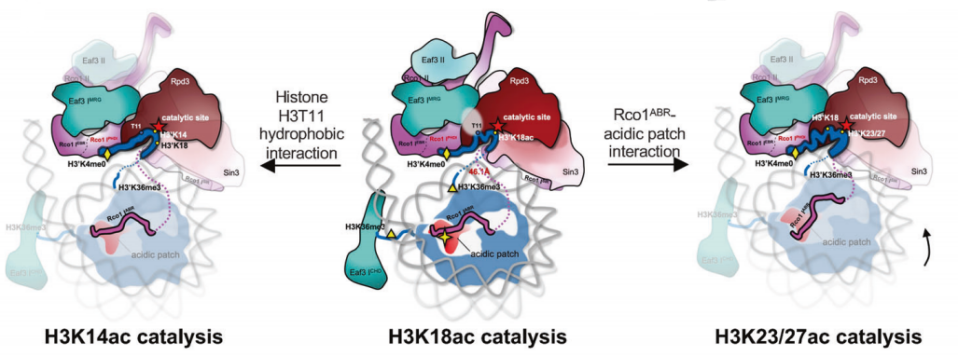
图2. Rpd3S如何选择和催化H3尾部多个特定赖氨酸残基的机制
该研究报道了Rpd3S全酶结合和催化核小体底物的结构基础,清晰揭示了Rpd3S全酶底物识别特异性和催化复杂性(图2),阐明了Rpd3S通过构象变化实现组蛋白多位点催化的机制,为研制高特异性Rpd3S/Sin3B抑制剂用于癌症治疗提供了新的靶点。
文章发表后,表观遗传领域的顶尖科学家Michael J. Carrozza 和 Jerry L. Workman在Cell Research进行了研究亮点的评论,评价这项工作为Rpd3S/HDAC结合核小体DNA和组蛋白尾巴的结合特性提供了新的见解,为其识别特定乙酰化赖氨酸的独特特性提供了引人入胜的视角 (https://www.nature.com/articles/s41422-023-00899-9)。
这项工作是在中国科学院战略先导B专项和国家自然科学基金项目的资助下完成的;中国科大生医部王雪娟教授和蔡刚教授为该论文的共同通讯作者,博士生张跃跃和博士后徐梦雪为该论文的共同第一作者,研究生王坡、周佳慧、王光显和韩帅龙参与了部分工作。数据收集在中国科学技术大学冷冻电镜中心完成。此外,该研究还得到了清华大学李海涛、陈柱成教授,中国科大臧建业、施蕴渝教授的大力帮助。
原文链接:https://www.nature.com/articles/s41422-023-00884-2
(生命科学与医学部、科研部)
